理化学研究所(理研)開拓研究所 杉田理論分子科学研究室の杉田 有治 主任研究員(計算科学研究センター 粒子系生物物理研究チーム チームプリンシパル)、本橋 昌大 大学院生リサーチ・アソシエイトらの共同研究グループは、生体内でエネルギー変換を担う分子モーター[1]である「F1-ATPase」の回転機構について、計算機シミュレーションを用いて解析し、原子レベルの「歪(ひず)み」と「押し出し」によって回転運動が生まれる仕組みを明らかにしました。
本研究成果は、細胞内で働く分子モーターの仕組みの理解を深め、生体ナノマシンの解明や人工分子モーターの設計、ナノテクノロジーへの応用に貢献すると期待されます。
共同研究グループは、スーパーコンピュータ「富岳」[2]と分子動力学ソフトウエア「GENESIS」[3]を用いて、好熱菌由来のF1-ATPaseにおける80度サブステップ回転を原子レベルで再現する分子動力学(MD)計算[4]を行いました。その結果、回転が「歪み」と「押し出し」から成る2段階の固定子(回転しない部分)の動きによって駆動されることが明らかになりました。
本研究は、科学雑誌『Proceedings of the National Academy of Sciences(PNAS)』オンライン版(8月12日付)に掲載されました。
背景
F1-ATPaseは、ATP(アデノシン三リン酸)[5]の加水分解によって得られる化学エネルギーを機械的な回転運動に変換する生体内の分子モーターの一種です。ミトコンドリアや細菌などに広く存在し、ATP合成酵素複合体(FoF1-ATP synthase)の一部として、生命活動に必要なATPの合成や分解に関与しています(図1A)。
F1-ATPaseは、中央に位置する回転子(γサブユニット[6])と、それを取り囲む固定子(三つのαサブユニットと三つのβサブユニット)から構成され、ATPの加水分解により回転子が回転することで、エネルギー変換が行われます(図1B)。好熱菌由来のF1-ATPaseでは、この回転運動は120度のステップで進行し、さらにその1ステップが80度と40度の二つのサブステップに分かれることが、単一分子観察実験などで示されています(図1C)。それぞれのサブステップ回転前の回転停止は、「ATP結合待ち」と「ATP加水分解待ち」と呼ばれます。最初の80度回転はATPの結合と密接に関係しており、エネルギー変換の駆動源として重要と考えられています。
これまで、F1-ATPaseの回転機構に関する研究は、単一分子実験や構造解析によって多くの知見が得られてきましたが、回転運動がどのように進行し、どのような分子間相互作用によって駆動されるのか、その詳細なメカニズムは十分に解明されていませんでした。
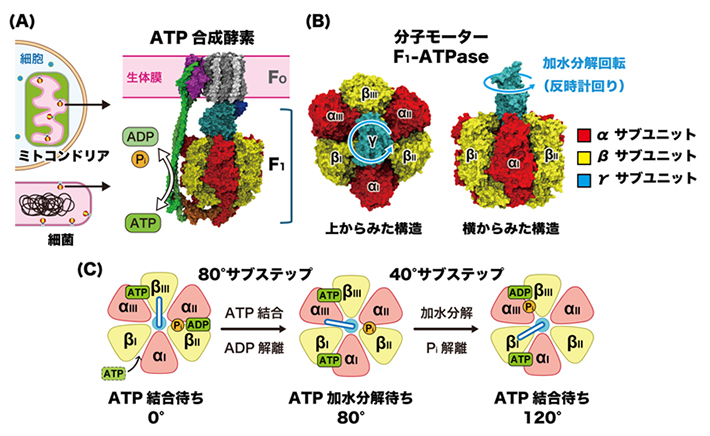
図1 ATP合成酵素とF1-ATPaseの構造、およびサブステップ回転の模式図
- (A)ATP合成酵素は、ミトコンドリア内膜(真核生物)や細胞膜(細菌)などの生体膜に存在する酵素複合体である。膜を貫通するFo部分と、膜の外側に露出した可溶性のF1部分から構成され、F1がATP合成を担う。
- (B)F1-ATPaseは、それぞれ三つのαサブユニットとβサブユニットが交互に配置された六量体(α3β3)と、その中央に位置する回転子γサブユニットから成り、これらで回転機能の最小単位を構成する。ATPの加水分解に伴い、γサブユニットは固定子であるα3β3複合体に対して反時計回りに回転する。
- (C)好熱菌由来のF1-ATPaseは、γサブユニットが80度と40度のサブステップ回転で構成された120度のステップ回転を行う。80度サブステップには、ATP結合とアデノシン二リン酸(ADP)解離が関与し、40度サブステップにはATP加水分解と無機リン酸(Pi)解離が関与することが知られている。
研究手法と成果
共同研究グループは、F1-ATPaseの回転メカニズムのうち、ATPの結合とアデノシン二リン酸(ADP)[5]の解離が関わる80度サブステップ回転に着目し、その分子機構を明らかにするため、分子動力学(MD)計算を行いました。
MD計算には、理研を中心に長年開発している分子動力学計算ソフトウエア「GENESIS」を使用しました。GENESISは、「富岳」などの大規模並列計算環境に最適化されており、本計算システムのような50万原子以上から成る巨大な分子系においても、高精度かつ高速な計算を可能にします。
まず、F1-ATPaseの80度回転前後の構造として、近年クライオ電子顕微鏡法[7]で明らかになった「ATP結合待ち」と「ATP加水分解待ち」に相当する回転角度を持つ構造を基に、両状態をつなぐ構造変化経路の候補をターゲットMD(TMD)[8]で探索しました。この探索では、ATPの結合やADPの解離に伴って生じる固定子の構造変化の順序を変えた三つの経路を仮定し、それぞれのケースで回転子がどのように回転するかを調べました(図2A)。その結果、「ATPの結合による構造変化に続いて、ADPの解離による構造変化が起こる(経路1)」という順序において、回転子が80度回転し得ることが分かりました(図2B)。
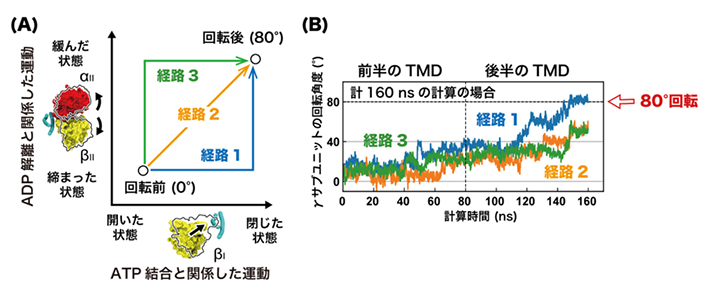
図2 ターゲットMDによる80度サブステップ回転の構造変化経路の探索
- (A)ターゲットMD(TMD)で探索した三つの構造変化経路の模式図。ATP結合やADP解離に伴う固定子の構造変化の順序を変えた三つの経路を仮定した。
- (B)TMDで誘起された固定子の構造変化に伴うγサブユニットの回転応答の時間変化。経路1においてγサブユニットが反時計回りに80度回転する動きが観察された。経路2および経路3では、反時計回りに回転したが80度には到達しなかった。ns:ナノ秒(nsは10億分の1秒)。
次に、80度回転した経路を基に、ストリング法[9]を用いて最も自然な構造遷移経路(最小自由エネルギー経路)を求めました。ストリング法では、構造の異なる複数のレプリカ構造(イメージ)を準備し、それぞれに対して並列にMDシミュレーションを行うことで、状態間を結ぶエネルギー的に最も妥当な経路を探索します。本研究では、64個のイメージを用いてストリング法を適用したため、通常のMD計算と比較して64倍多い計算量が必要でしたが、「富岳」上で効率の良いMD計算が可能なGENESISを用いたことで、このような大規模計算を実現することができました。
その結果、F1-ATPaseの80度回転は、(1)固定子の構造歪みによって回転が始まり、(2)回転子が周囲の構造から押し出されるようにして回転が完了する、という2段階の過程で駆動されていることが明らかになりました(図3)。このメカニズムは、従来提案されていた静電相互作用[10]や立体障害[11]による駆動力を包含するモデルとして、「歪み・押し出し機構(distortion-push mechanism)」と名付けられました。
本研究は、従来の単一構造解析や外力による回転シミュレーションでは見えなかった自然な回転駆動の全体像を明らかにしました。それは、生体分子モーターの力学的理解に大きな進展をもたらす成果といえます。
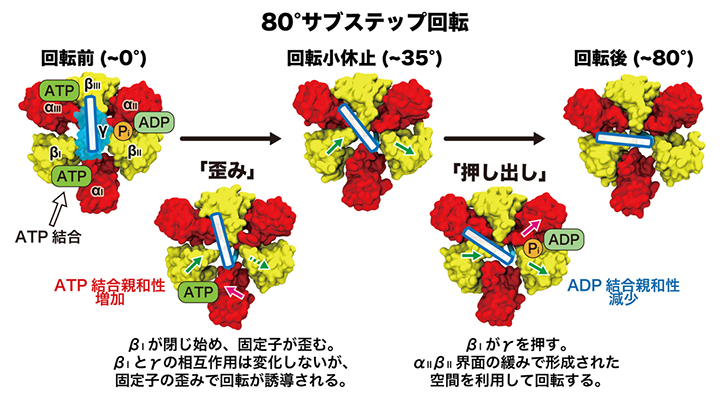
図3 生体分子モーターF1-ATPaseにおける80度サブステップの回転駆動機構
今回の研究で明らかにした、好熱菌由来のF1-ATPaseにおける80度サブステップの回転駆動機構。ATPの結合に伴い、βIが閉じ始めることで固定子に歪みが生じ、これが回転の開始を誘導する。この段階ではβIとγサブユニット(青色で囲った長方形)との相互作用は変化しない。続いて、βIがγサブユニットを直接押し出すように働き、αIIβII界面の緩みにより生じた空間を利用することで、γサブユニットが80度回転する。
今後の期待
F1-ATPaseを含むATP合成酵素は、細胞内で化学エネルギー(ATP)を合成・分解し、生命活動に必要なエネルギー変換を担う分子モーターです。これらの分子装置は生物種を超えて高度に保存されており、構造や機能の基本原理には共通性があります。そのため、本研究で明らかになったF1-ATPaseの回転駆動メカニズムは、他のATP駆動型分子モーターにも共通する普遍的な原理を含んでいる可能性があります。
また、F1-ATPaseのようなナノスケールのエネルギー変換機構を理解することは、生命科学の基礎研究として重要であると同時に、人工分子モーターの開発や、分子レベルでの機械的な動作を制御するナノテクノロジーへの応用にもつながると期待されます。
その実現には、今後も「富岳」などの大規模スーパーコンピュータと最先端の分子シミュレーション手法を組み合わせた、高精度な計算科学研究を継続的に進めていくことが重要です。
補足説明
- 1.分子モーター
化学エネルギーを使って、自ら運動を生み出す分子である。細胞内では、物質の輸送や細胞骨格の再構成、運動など、さまざまな生命現象に関与している。 - 2.スーパーコンピュータ「富岳」
スーパーコンピュータ「京」の後継機として理化学研究所が設置し、2021年3月から共用を開始した計算機。2025年6月のGraph500ランキングで11期連続1位を獲得。また、TOP500では7位、HPCG(High Performance Conjugate Gradient)では2位、HPL-AI Mixed Precision(HPL-MxP、旧名HPL-AI)では6位を獲得するなど、世界トップレベルの性能を持つ。 - 3.分子動力学ソフトウエア「GENESIS」
GENESISは、タンパク質や核酸、脂質、糖鎖などの生体高分子を対象とした分子動力学計算ソフトウエア。大規模並列計算や構造変化の探索、複数のスケールに対応した解析が可能であり、フリーソフトウエアとして公開されている。 - 4.分子動力学(MD)計算
タンパク質や核酸などの生体分子の構造や運動を、物理法則に基づいてコンピュータ上で再現するシミュレーション手法。分子に働く力を基に時間発展を追うことで、構造変化や分子間相互作用の過程を明らかにできる。MDはMolecular Dynamicsの略。 - 5.アデノシン三リン酸(ATP)、アデノシン二リン酸(ADP)
ATPは、生物全般に存在するリン酸化合物の一つであり、三つのリン酸が結合している。ATPが加水分解されてリン酸が一つ外れるとADPとなる。この反応で放出されるエネルギーは、さまざまな生命活動に利用されることから、ATPは「エネルギーの通貨」とも呼ばれている。 - 6.サブユニット
複数のタンパク質が集合してできた複合体における構成要素の一つ。 - 7.クライオ電子顕微鏡法
試料を急速に凍結して観察する電子顕微鏡技術であり、生体分子を結晶化せずに高分解能で構造解析することが可能である。 - 8.ターゲットMD(TMD)
シミュレーション対象の分子をある特定の構造へと誘導しながら、その過程を解析する分子動力学手法。初期構造から目標構造までの構造変化の経路を探索するのに有効である。TMDはtargeted MDの略。 - 9.ストリング法
初期構造と最終構造をつなぐ反応経路を、エネルギー的に最も自然な形で計算する手法。複数の構造点(イメージ)をひも状につなぎ、最小エネルギー経路を求めることで、分子の構造変化の過程や遷移状態を明らかにする。 - 10.静電相互作用
正と負の電荷を持つ原子や分子の間に働く引力または斥力。生体分子間では、アミノ酸残基の電荷によってタンパク質の立体構造や分子間の結合状態が影響を受ける。 - 11.立体障害
原子や分子が物理的に近づき過ぎることで空間的な衝突や干渉が生じ、自由な構造変化や結合を妨げる現象。分子の構造や反応性、結合様式に制限を与える要因として働く。
共同研究グループ
理化学研究所
開拓研究所 杉田理論分子科学研究室
主任研究員 杉田 有治(スギタ・ユウジ)
(計算科学研究センター 粒子系生物物理研究チーム チームプリンシパル)
大学院生リサーチ・アソシエイト 本橋 昌大(モトハシ・マサヒロ)
(中央大学 大学院理工学研究科 物理学専攻 博士課程3年)
基礎科学特別研究員(研究当時)大出 真央(オオイデ・マオ)
(現 理研 開拓研究所 杉田理論分子科学研究室 客員研究員、大阪大学 蛋白質研究所 助教、科学技術振興機構さきがけ研究者)
計算科学研究センター 粒子系生物物理研究チーム
上級技師 小林 千草(コバヤシ・チグサ)
研究員 ジョン・ジェウン(Jung Jaewoon)
(開拓研究所 杉田理論分子科学研究室 専任技師)
中央大学 大学院理工学研究科 物理学専攻
教授 宗行 英朗(ムネユキ・エイロウ)
研究支援
本研究は、理化学研究所大学院生リサーチ・アソシエイト制度(研究者:本橋昌大)、同基礎科学特別研究員制度(研究者:大出真央)、および理化学研究所運営費交付金(独創的研究課題「細胞内環境の生物学(課題代表:杉田有治)」「糖と脂質の構成原理を読み解く先端研究(課題代表:杉田有治)」、同奨励課題「分子動力学計算での高効率な解析手法の開発(課題代表:ジョン・ジェウン)」)、日本学術振興会(JSPS)科学研究費助成事業基盤研究(S)「マルチスケール分子動力学シミュレーションによる細胞内分子動態の解明(研究代表者:杉田有治、19H05645)」、同学術変革領域研究(A)「クロススケール細胞内分子構造動態の実験データ融合モデリング(研究代表者:杉田有治、21H05249)」「非ドメイン型バイオポリマーの分子動力学計算(研究代表者:依田隆夫、21H05282)」、文部科学省データ創出・活用型マテリアル研究開発プロジェクト事業「バイオ・高分子ビッグデータ駆動による完全循環型バイオアダプティブ材料の創出(研究代表者:沼田圭司、JPMXP1122714694)」、同スーパーコンピュータ「富岳」成果創出加速プログラム「計算材料科学が主導するデータ駆動型研究手法の開発とマテリアル革新(課題代表者:久保百司、PMXP1020230327)」、科学技術振興機構(JST)戦略的創造研究推進事業個人型研究(さきがけ)「細胞の動的高次構造体(研究総括:野地博行)」の研究課題「実験と計算の協奏による生体分子動態解析法の開発(研究者:大出真央、JPMJPR22E2)」による助成を受けました。また、分子動力学シミュレーションは、HPCIシステム利用研究課題(課題番号:hp230111、hp240047)を通じたスーパーコンピュータ「富岳」の計算資源、「富岳」高度化・利用拡大枠(課題番号:ra240003)ならびに理化学研究所計算科学研究センターのスーパーコンピュータ「HOKUSAI BigWaterfall」(プロジェクト番号:Q22535、Q22595)の計算資源の提供を受け、実施しました。
原論文情報
- Masahiro Motohashi, Mao Oide, Chigusa Kobayashi, Jaewoon Jung, Eiro Muneyuki, and Yuji Sugita, "The distortion-push mechanism for the γ-subunit rotation in F1-ATPase", Proceedings of the National Academy of Sciences, 10.1073/pnas.2502642122
発表者
理化学研究所
開拓研究所 杉田理論分子科学研究室
主任研究員 杉田 有治(スギタ・ユウジ)
(計算科学研究センター 粒子系生物物理研究チーム チームプリンシパル)
大学院生リサーチ・アソシエイト 本橋 昌大(モトハシ・マサヒロ)
発表者のコメント
分子動力学シミュレーションを通じてF1-ATPaseのダイナミックな回転運動を可視化できたことを、うれしく思います。また、本研究を支えてくださった共同研究者の皆さまに心より感謝いたします。(本橋 昌大)
この研究は、私が理研で研究室を主宰する以前(2007年以前)から、本橋君の指導教官の宗行教授と夢見ていたものでした。本橋君の頑張りと、「GENESIS」や「富岳」などのソフトウエア・ハードウエアの革新によって実現できたことは大変うれしいです。残りの40度回転も含めて、分子モーターの回転機構の完全な理解を目指します。(杉田 有治)
報道担当
理化学研究所 広報部 報道担当
お問い合わせフォーム
