理化学研究所(理研)生命医科学研究センター 免疫器官形成研究チームの古関 明彦 チームディレクター、近藤 隆 上級研究員、伊藤 伸介 研究員らの共同研究グループは、遺伝子のオン・オフの切り換えを管理しているポリコム複合体[1]の遺伝子発現調節メカニズムの一端を明らかにしました。
本研究成果は、ES細胞(胚性幹細胞)[2]やiPS細胞(人工多能性幹細胞)[2]の細胞分化コントロールが必要な再生医療やがんや疾患などの理解につながり、治療法開発の基礎的知見になると期待されます。
今回、共同研究グループは、ポリコム複合体の一つであるPCGF1-PRC1を構成する因子の一つ、SKP1Aを欠損させたES細胞ならびにマウス胎児の解析を行い、ポリコム複合体PCGF1-PRC1がPRC2[1]の構成因子EEDをユビキチン化[3]し、タンパク質分解酵素による分解を誘導することで、細胞分化時において、それまで抑制状態にあったポリコム標的遺伝子群の転写活性化に寄与することを発見しました。
本研究は、科学雑誌『Molecular Cell』(9月4日号)の掲載に先立ち、オンライン版(8月25日付)に掲載されました。
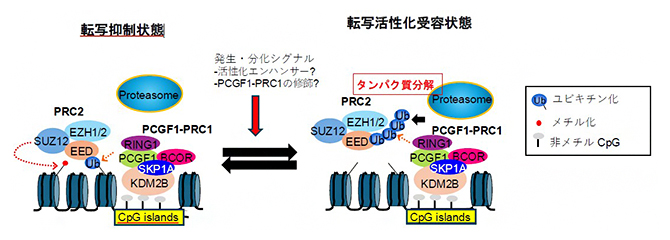
本研究で明らかにしたポリコム複合体PCGF1-PRC1による転写抑制解除機構の概要
背景
生命の発生・分化過程において、ゲノムDNAのユークロマチン領域[4]に存在する遺伝子の正確な転写調節は正常な個体発生、細胞の運命決定に必須です。遺伝子の働きは、DNAやそれと結合するヒストンタンパク質[5]の化学修飾によって制御されており、それをエピジェネティックス[6]による制御と呼びます。細胞の状態が変化する際に、必要な遺伝子の転写は活性化され、不要な遺伝子は抑制状態に移行します。ポリコム複合体は遺伝子の転写抑制に重要な役割を担う因子の一つとして知られています。
ポリコム複合体は構成因子としてヒストン修飾酵素を含んでいることが知られています。発生および細胞分化に関連する重要な遺伝子の多くは、ゲノムDNA上のプロモーター領域[7]にCpGアイランド[8]を有しています。このCpGアイランドにポリコム複合体が集積することで、ヒストンの翻訳後修飾などのエピジェネテックな修飾を介して下流の遺伝子の転写が抑制される考えられています。ポリコム複合体が集積する標的遺伝子群は転写活性化に際して、ポリコム複合体をプロモーターから除去する必要がありますが、その除去過程、除去の機構については全く知られていませんでした。
そこで共同研究グループはロモーターからポリコム複合体を除去する機構の解明に挑みました。
研究手法と成果
ポリコム複合体は、複合体を構成する因子の違いにより大きく分けてPRC1[1]、PRC2の二種類に区分されます。しかし、近年の研究によりPRC1はさらに構成因子の異なる6種類のサブタイプ(PCGF1-PRC1、PCGF2-PRC1、PCGF3-PRC1、PCGF4-PRC1、PCGF5-PRC1、PCGF6-PRC1)に分けられることが明らかになりました。
これらの複合体はそれぞれ特徴的なPCGF因子(PCGF1からPCGF6)によって構成されており、PRC1の共通因子としてユビキチン化酵素E3の構成要素であるRING1A/Bを有しています。PRC1は、ヒストンH2A[5]の119番目のリジンにモノユビキチン(H2AK119ub1)を付与することによって、遺伝子の発現が抑制されることが知られています。中でもPCGF1-PRC1は、RING1A/Bの他に、SKP1Aというもう一つのユビキチン化酵素の構成因子を含んでいることが分かっており、ヒストンのモノユビキチン化以外のユビキチン化に関与している可能性が示唆されていました。
共同研究グループは、マウスES細胞(胚性幹細胞)、およびその分化、さらにはマウス胎児の発生時におけるSKP1Aの役割について明確にするため、Skp1a遺伝子を欠損させたES細胞やマウスなどを用いて、プロテアソーム[9]阻害剤投与群との比較検討を行いました。
はじめにPCGF1-PRC1がモノユビキチン化以外の活性を有しているかどうかを検証しました。ES細胞からPCGF1-PRC1特異性構成因子であるKDM2Bと相互作用するタンパク質を精製して、プロテオーム解析[10]を行いました。ES細胞はプロテアソーム阻害剤MG132で前処理し、プロテアソームによる急速な分解によって失われる可能性のあるKDM2B相互作用タンパク質の捕捉を試みました。未処理ES細胞から精製したタンパク質複合体は、PCGF1-PRC1が主な相互作用因子として検出されました。一方で、MG132処理ES細胞から精製したタンパク質複合体は、PCGF1-PRC1に加えて、プロテアソームの多くの構成因子、PRC2構成因子が検出されました(図1A)。さらに、精製したタンパク質複合体にはポリユビキチン化タンパク質も濃縮されていることが示されました。これらの結果から、PCGF1-PRC1がユビキチン-プロテアソーム系に関与すること、PRC2との相互作用がプロテアソーム活性により制御されていることが示唆されました。
次に、ES細胞におけるPCGF1-PRC1、SKP1A、プロテアソーム(PSMC5)、ポリユビキチン鎖の分布を明らかにするために、クロマチン免疫沈降シーケンス(ChIP-seq)[11]を行いました。その結果、PCGF1-PRC1のみならず、SKP1A、PSMC5、ポリユビキチン鎖のCpGアイランドへの強い濃縮が検出されました (図1B)。また、SKP1A遺伝子欠損ES細胞において、ポリユビキチン鎖のシグナルが大幅に低下しました。この結果から、PCGF1-PRC1と結合するSKP1AがCpGアイランド上でポリユビキチン化を行い、プロテアソームによるタンパク質分解を誘発することが示唆されました。
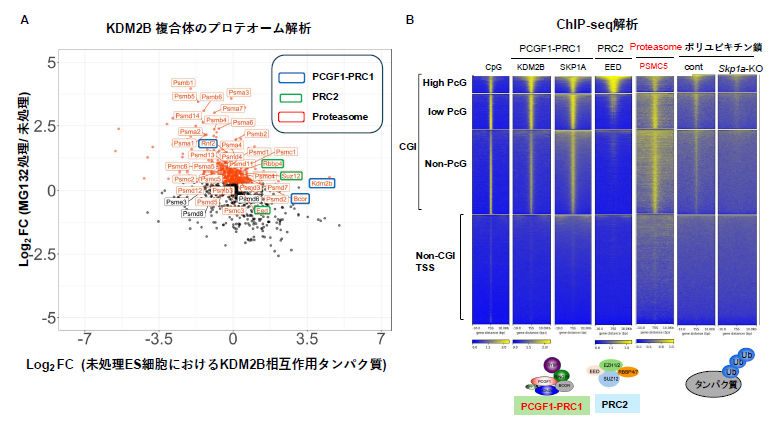
図1 KDM2B複合体のプロテオーム解析とCpGアイランドにおける局在
(A)KDM2B複合体のプロテオーム解析。MG132処理によりプロテアソーム(Proteasome)との相互作用が増強される。(B)ChIP-seq解析によりPCGF1-PRC1、プロテアソームがCpGアイランドに局在することが観察された。MG132処理した野生型(cont)、Skp1a-欠損(KO)ES細胞を用いてポリユビキチン化タンパク質のChIP-seq解析を行った結果、Skp1a-KO ES細胞ではポリユビキチン化タンパク質がCpGアイランドから消失することが観察された。
また、SKP1Aによるタンパク質分解の標的を探索するためにES細胞でのSkp1a欠損、タンパク質分解酵素阻害剤投与を用いた、ユビキチン化タンパク質解析(ユビキチローム)を行いました。その結果、SKP1Aを含むPCGF1-PRC1によりユビキチン化されタンパク質分解の基質となる標的の一つとしてPRC2の構成因子であるEEDを同定しました。
遺伝子群でのポリコム複合体の集積の変化について、次世代シーケンサー[12]を用いたChIP-seqおよびCUT & Tag法[13]による観測により、Skp1a欠損、タンパク質分解酵素阻害剤投与においてはポリコム複合体の蓄積が上昇すること、また、それに伴って、マウスの発生におけるMeis2[14]の遺伝子発現が抑制されることを観察しました(図2)。
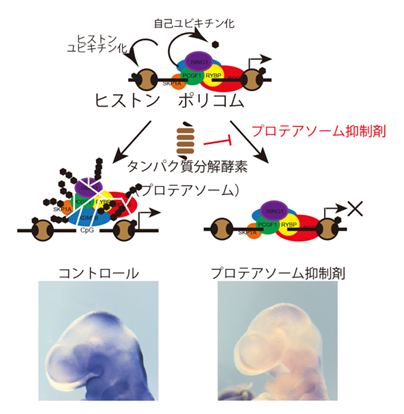
図2 ポリコム複合体蓄積による遺伝子発現の抑制
タンパク質分解を抑制するとポリコム複合体が蓄積し、発生により誘導されるMeis2遺伝子の発現が生じず、抑制状態が継続する。青く染まった領域は、Meis2遺伝子が発現していることを示す。
本研究により、CpGアイランドがプロテアソーム結合の足場になっており、そこに備わっているPCGF1-PRC1とSKP1Aを介したユビキチン化が遺伝子発現制御の一翼を担っていることが明らかになりました。また、PRC2の構成因子EEDをユビキチン化の標的として見いだし、PRC2の分解が標的遺伝子群の転写活性化につながることを発見しました。
今後の期待
遺伝子の発現調節メカニズムの解明は、形態形成だけでなく、細胞の正常な分化・脱分化の過程の解明など、発生に関わるさまざまな研究において重要視されています。
今後、さらにポリコム複合体による遺伝子発現制御の詳細な仕組みを明らかにし、遺伝子発現のオン・オフの切り換えの核心に迫ることで、ES細胞やiPS細胞の細胞分化のコントロールが必要な再生医療やがんや疾患などの理解につながり、治療法開発の基礎的知見になると期待されます。
補足説明
- 1.ポリコム複合体、PRC2、PRC1
ポリコム複合体は、複数のポリコムタンパク質が集合した複合体。ポリコム複合体は大きく分けてPRC1とPRC2の2種類に区分される。PRC1はヒストンH2A([5]参照)の119番目のアミノ酸残基であるリジンのモノユビキチン化(H2AK119ub1)、PRC2はヒストンH3の27番目のアミノ酸残基であるリジンのメチル化(H3K27me3)とそれぞれ抑制性のヒストン修飾を行う。PRC2は、EED、SUZ12、EZH1/2のコアサブユニットから構成される。 - 2.ES細胞(胚性幹細胞)、iPS細胞(人工多能性幹細胞)
ES細胞は、受精卵が分化してできた発生初期の胚の細胞からつくられ、全ての細胞に分化する能力を持つ。iPS細胞は、体細胞に数種類の転写因子を導入することにより、「分化万能性」を持たせた細胞。 - 3.ユビキチン化
ユビキチン修飾系によってエネルギー依存的に特定のタンパク質にユビキチンが結合する反応。 - 4.ユークロマチン領域
遺伝子がより多く含まれているクロマチンの形状または種類のことで、転写が頻繁な領域である。 - 5.ヒストンタンパク質、ヒストンH2A
ヒストンタンパク質とは、ゲノムDNAを折り畳んで核内に収納する役割を持つ。主要なヒストンタンパク質はH2A、H2B、H3、H4の4種類に分類される。それぞれ2分子ずつ集まり、ヒストン8量体(ヒストンオクタマー)を形成する。一つのヒストン8量体は、約146bp(塩基対)のDNAを左巻きに約1.65回巻き付け、ヌクレオソームを構築する。ヌクレオソームはクロマチン構造の最小単位。 - 6.エピジェネティクス
先天的に遺伝するDNA配列によるものではなく、後天的な遺伝子発現制御メカニズム(DNAのメチル化やヒストン修飾など)の総称。 - 7.プロモーター領域
ゲノムDNA上でRNAに書き写される領域の近くにあり、遺伝子を発現させる機能を持つ部分。 - 8.CpGアイランド
ゲノムにおいて、シトシン(C)の次にグアニン(G)という順番で並ぶ2塩基配列(CpG)の頻度が高い領域。哺乳類のプロモーターの多くに見られる。脊椎動物ではCpGのCの5位炭素が高頻度でメチル化されているが,CpGアイランドは一般的に低メチル状態である。 - 9.プロテアソーム
タンパク質の分解を行う巨大な酵素複合体。ユビキチン分子が複数連結されたタンパク質の多くは、プロテアソームによって分解を受ける。ユビキチン-プロテアソーム系によるタンパク質分解は細胞周期制御、シグナル伝達といった多くの生物学的プロセスの制御に関わっている。 - 10.プロテオーム解析
生物の持つタンパク質の発現量や修飾状態を網羅的に解析する手法。 - 11.クロマチン免疫沈降シーケンス(ChIP-seq)
クロマチン免疫沈降法は、生体内におけるタンパク質とゲノムDNAの結合部位を検出するための実験手法である。ホルムアルデヒドによってDNAとDNAに結合しているタンパク質を架橋した後に、DNAの断片化を行い、タンパク質の抗体を用いてタンパク質-DNA複合体を回収する。さらに、DNAとタンパク質の架橋を解いてDNAのみを回収し、その配列を調べることでタンパク質がどのようなDNA配列に結合していたかが分かる。次世代シーケンスと組み合わせることで、ゲノム上のどこに、どれくらいの強さで結合していたかが分かる。ChIP-seqはChromatin Immunoprecipitation sequencerの略。 - 12.次世代シーケンサー
DNAやRNAの塩基配列を高速で解読する装置であり、大量のデータを効率的に取得できる。 - 13.CUT & Tag法
ChIP-seqの代替となる、より迅速で低コストな解析方法。CUT & Tagは、ChIP-seqに比べてより少ないサンプル量でクロマチンをプロファイリングできる。CUT & TagはCleavage Under Targets & Tagmentationの略。 - 14.Meis2
白血病の原因遺伝子の一つとして発見された。ショウジョウバエにおけるホメオティック遺伝子群(Hox)のコファクターとして知られるhomothoraxのホモログ(相同体)であり、TALE(three amino acid loop extension)ホメオボックス遺伝子で、転写調節因子の一つでもある。近年、Hox遺伝子群以外の多くの転写調節因子(HLH、Znフィンガーなど)とも複合体を形成することが判明した。哺乳動物にはMeis1、2、3が存在する。Meisはmyeloid ecotropic insertion siteの略。
共同研究グループ
理化学研究所 生命医科学研究センター
免疫器官形成研究チーム
チームディレクター 古関 明彦(コセキ・ハルヒコ)
上級研究員 近藤 隆(コンドウ・タカシ)
研究員 伊藤 伸介(イトウ・シンスケ)
研究員 高野 淳一朗(タカノ・ジュンイチロウ)
特別研究員(研究当時)椙下 紘貴(スギシタ・ヒロキ)
(現 東京大学 大学院薬学系研究科 薬科学専攻生物薬科学講座 特任助教)
専任研究員 ジャファル・シャリフ(Jafar Sharif)
研究員 原地 美緒(ハラチ・ミオ)
テクニカルスタッフⅡ 柴田(石井)真利花(シバタ・マリカ)
研究パートタイマーⅠ 近藤 香(コンドウ・カオリ)
統合ゲノミクス研究チーム
研究員(研究当時) ヨンウン・ハン(Yong-Woon Han)
(現 名古屋市立大学大学院医学研究科 分子腫瘍学分野 講師)
技師 遠藤 高帆(エンドウ・タカホ)
チームディレクター 清田 純(セイタ・ジュン)
かずさDNA研究所
応用プロテオミクスグループ
グループ長 川島 祐介(カワシマ・ユウスケ)
オミックス解析施設
施設長 小原 收(オハラ・オサム)
オミックス医科学研究室
特任研究員 中山 学(ナカヤマ・マナブ)
研究支援
本研究は、日本医療研究開発機構(AMED)革新的先端研究開発支援事業ユニットタイプ「エピゲノム研究に基づく診断・治療へ向けた新技術の創出(研究総括:山本雅之)」研究領域における研究課題「エピジェネティクスによるエンハンサー動態制御メカニズムの解明と細胞機能制御への応用(研究代表者:古関明彦)」、同ムーンショット型研究開発等事業「細胞運命転換を用いた若返りによるがんリスク0の世界(研究開発代表者:古関明彦、JP22zf0127008)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究(A)「ポリコム群抑制クロマチン形成におけるCpG配列認識の意義の解明(研究代表者:古関明彦)」、同基盤研究(C)「クロマチン制御複合体PRC1がもつポリユビキチン化活性が制御する遺伝子発現動態の解明(研究代表者:伊藤伸介)」、内藤記念科学奨励金・若手ステップアップ研究助成「CpGアイランドのエピゲノム制御機構の解明(研究代表者:伊藤伸介)」による助成を受けて行われました。
原論文情報
- Takashi Kondo, Shinsuke Ito, Junichiro Takano, Yong-Woon Han, Takaho Endo, Kaori Kondo, Marika Shibata, Yusuke Kawashima, Hiroki Sugishita, Mio Harachi, Jafar Sharif, Jun Seita, Osamu Ohara, Manabu Nakayama, and Haruhiko Koseki, "SKP1A bound to Polycomb-silenced genes mediates degradation of PRC2 and preconditions their activation", Molecular Cell, 10.1016/j.molcel.2025.08.004
発表者
理化学研究所
生命医科学研究センター 免疫器官形成研究チーム
チームディレクター 古関 明彦(コセキ・ハルヒコ)
上級研究員 近藤 隆(コンドウ・タカシ)
研究員 伊藤 伸介(イトウ・シンスケ)
 古関 明彦
古関 明彦
 近藤 隆
近藤 隆
 伊藤 伸介
伊藤 伸介
報道担当
理化学研究所 広報部 報道担当
お問い合わせフォーム
